Simulateur d'évolution d'épidémie de Virus, Covid-19 ou autre, via un modèle compartimental
Ce simulateur montre une progression selon un modèle SIR étendu
(Sain / Exposé / Infecté / Guéri (Recovered) / Hospitalisé / Décédé).
Vous pouvez faire varier les paramètres sous le simulateur et modifier manuellement la courbe R0 directement sur le graph (pour simuler des vagues de confinement/déconfinement par exemple).
A et C représente respectivement l'arrêt d'import des cas et la mise en place du confinement.
Tout échange et tout retour de terrain (notamment en Polynésie Française) sur la paramétrisation du modèle seront les bienvenus.
Par défaut, le 1er jour du graph est à 100 cas infectés (+50 exposés). Ceci correspond pour la Polynésie au 15 août. On peut bien sûr modifier cela.
Par défaut également, la série R est initialisée avec les valeurs communiquées jusqu'au 22/11/2020 en PF et ensuite R prend une valeur constante à 1.15 (hypothèse optimiste avec toutes mesures inchangées).
Enfin, vous pouvez modifier R(t) sur toute la série. Pour ce faire, afficher R0 et vous pourrez modifier par drag and drop sur chaque point (fastidieux et un export, modification puis import est peut être plus simple).
Attention, si vous changez la largeur temporelle du graph, il faudra recommencer, il est donc conseillé d'exporter R(t) si vous l'avez modifiée...
Vous pouvez modifier les paramètres dans chaque groupe ci-dessous :
Aspects techniques
Schémas de principe
Schématiquement, les compartiments se présentent comme suit :
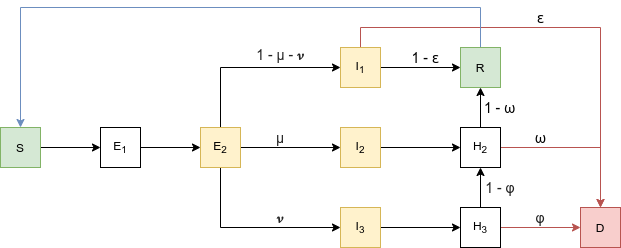
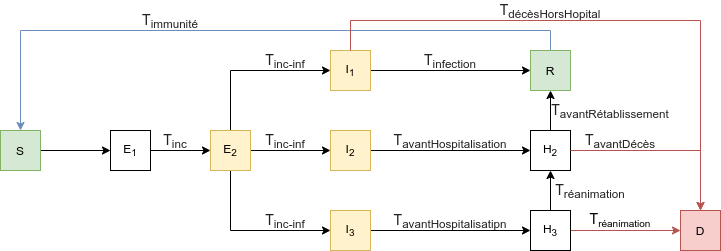
Les différents niveaux de soins sont représentés par H2 et H3, la différenciation se fait en amont (I1, I2, I3) afin d'avoir une différentation des durées (si besoin) et de pouvoir appliquer des statistiques connues.
Dans les paramètres d'initialisations, on prend comme hypothèse I1(0) = Nombre d'infectés initiaux, et E(0) = 0.5 * I1(0)
Définitions
Jeu d'équations utilisées
NB : on utilise les populations normalisées et I désigne par exemple la proportion de personnes infectées dans la population totale.
R0 et taux de transmission
Dans un modèle SIR simple, on peut écrire :
Dans le présent modèle, on aura :
où βe est le taux de transmission dans un état infectieux e, p(e) la probabilité de cet état et Te la durée de cet état. On utilise en outre un seul taux β représentant le taux de transmission dans l'état I1 et des coefficients αe pour ajuster les autres taux. Ainsi exprimé, on aura :
Initialisation des paramètres (cas du Covid-19/SARS-CoV-2)
Littérature
Les valeurs ne sont pas établies aujourd'hui avec certitudes et la littérature récente donne plusieurs valeurs pour les différentes caractéristiques de la maladie. En voici qui ont servi ici :
| Source | Paramètre | Valeur |
|---|---|---|
| https://www.imperial.ac.uk/media/imperial-college/medicine/sph/ide/gida-fellowships/Imperial-College-COVID19-Europe-estimates-and-NPI-impact-30-03-2020.pdf | R0 | 3.87 (sans confinement) / 1.43 (avec confinement) |
| https://wwwnc.cdc.gov/eid/article/26/7/20-0282_article | R0 | 5.7 |
| https://wwwnc.cdc.gov/eid/article/26/7/20-0282_article (table 5) | TContagion | 9 jours |
| https://wwwnc.cdc.gov/eid/article/26/7/20-0282_article | TIncubation | 5.2 jours |
| https://wwwnc.cdc.gov/eid/article/26/7/20-0282_article | TAvantHospitalisation | 5.5 jours (hors épidémie surveillée) / 1.5 jours (en épidémie surveillée) |
| https://wwwnc.cdc.gov/eid/article/26/7/20-0282_article | TAvantRétablissement | 11.5 jours |
| https://wwwnc.cdc.gov/eid/article/26/7/20-0282_article | TRéanimation | 11.2 jours (avant décès) |
| https://www.thelancet.com/journals/lancet/article/PIIS0140-6736(20)30566-3/fulltext | TContagion | 20 jours |
| https://annals.org/aim/fullarticle/2762808/incubation-period-coronavirus-disease-2019-covid-19-from-publicly-reported?searchresult=1 | TIncubation | 5.1 jours |
Fitting
Une fois les valeurs préssentie assignée, il convient de modifier certains paramètres dépendant de la population ou des équipements sanitaires : R0 ainsi que les paramètres de suites cliniques.
Idéalement, il faudrait appliquer une minimisation des écarts sur une série de données connue.
Limitations, améliorations
Par défaut, on fait l'hypothèse que les patients guéris sont immunisés mais vous pouvez activer une immunité temporaire dans les paramètres techniques.
On ne tient pas compte de la capacité de charge hostpitalière, on peut imaginer que le taux de décès soit une fonction de celle ci, et introduire une transition H2 vers H3 dépendant de la capacité hospitalière également.
Une propagation 2D pourrait être représentée par ce modèle en considérant des compartiments par zones géographiques (pour les I et E essentiellement) et des paramètres par zone, tenant compte de la densité de population notamment, ainsi que des flux d'échanges.
De la même façon, une décomposition en compartiments d'âge serait possible, moins pour visualiser les effets par tranche d'âge que pour mieux modéliser les transmissions. Il faudrait revoir les équations pour généraliser en utilisant des matrices.
Hypothèse de population constante : on ne tient pas compte de la mortalité naturelle ni de la natalité ni des migrations de population entre le système et l'extérieur. Si on voulait en tenir compte (sur une période longue), il suffirait d'ajouter des taux à S et D, en admettant que les entrants sont sains.
Références
Pour le principe
- Fondation de la théorie des modèles à compartiments : https://royalsocietypublishing.org/doi/pdf/10.1098/rspa.1927.0118
- https://interstices.info/modeliser-la-propagation-dune-epidemie/
- https://fr.wikipedia.org/wiki/Mod%C3%A8les_compartimentaux_en_%C3%A9pid%C3%A9miologie
- http://www.math.ens.fr/enseignement/telecharger_fichier.php?fichier=1693
- http://marcchoisy.free.fr/pdf/DeBoeck2010.pdf
- https://www.theses.fr/2012PA05S010.pdf
- https://web.stanford.edu/~jhj1/teachingdocs/Jones-on-R0.pdf
- Sur la séparation des états en incubation : https://hal-pasteur.archives-ouvertes.fr/pasteur-02548181/document
Pour les initialisations des paramètres
- Sur R0 particulièrement : https://www.imperial.ac.uk/media/imperial-college/medicine/sph/ide/gida-fellowships/Imperial-College-COVID19-Europe-estimates-and-NPI-impact-30-03-2020.pdf
- Sur R0, Tinc, TavantHosp, TavantRet : https://wwwnc.cdc.gov/eid/article/26/7/20-0282_article
- https://www.infectiologie.com/fr/actualites/covid-19-nouveau-coronavirus_-n.html
- Sur la durée d'inbation : https://annals.org/aim/fullarticle/2762808/incubation-period-coronavirus-disease-2019-covid-19-from-publicly-reported?searchresult=1
- Sur la durée de contagion : https://www.ecdc.europa.eu/en/covid-19/questions-answers
- Stats Islande (population : 360.000 habs en 2020) : https://www.covid.is/data
- Sur la durée d'immunité : https://www.gov.uk/government/publications/react-2-real-time-assessment-of-community-transmission-antibody-waning/react-2-real-time-assessment-of-community-transmission-antibody-waning
Historique
- Mars 2020 : Création
- Avril 2020 : Séparation Etats E
- Avril 2020 : Ajout exports/imports
- 20 octobre 2020 : Actualisation paramètres initiaux PF
- 04 novembre 2020 : Actualisation paramètres initiaux PF
- 04 novembre 2020 : Ajout de la réinfection possible
- 04 novembre 2020 : Ajout de la réinfection possible
- 23 novembre 2020 : Actualisation courbe R0 avec suite à 1.3
- 30 novembre 2020 : Actualisation courbe R0 avec suite optimiste à 1.15